Introducción
Dentro del género Mycoplasma, existen hemoplasmas que infectan humanos (Candidatus Mycoplasma haemohominis) y a diversos animales, incluyendo al ganado bovino (Candidatus Mycoplasma haemobos, Candidatus Mycoplasma haemocervae y Mycoplasma wenyonii), gatos y perros (Mycoplasma haemocanis y Mycoplasma haemofelis), ratas y ratones (Mycoplasma coccoides, Candidatus Mycoplasma haemomuris), monos (Candidatus Mycoplasma haemomacaque), llamas y alpacas (Candidatus Mycoplasma haemolamae), ciervos (Candidatus Mycoplasma erythrocervae) y ovejas (Mycoplasma ovis y Candidatus Mycoplasma haemovis) (Messick et al. 2002; Hackett et al. 2006; Girotto et al. 2012; do Nascimento et al. 2012; Conrado et al. 2015; Suksai et al. 2016).
El hemoplasma Ca. M. haemobos es considerado como un agente causal de anemia infecciosa en mamíferos al adherirse a los eritrocitos del hospedero (Girotto et al. 2012). Es una bacteria Gram negativa no cultivable que carece de pared celular, cuya transmisión mecánica en el ganado ocurre por medio de vectores como moscas (Stomoxys calcitrans (L.)), mosquitos (Aedes y Culex spp.), tábanos (Haematopota pluvialis L. y Tabanus spp.) y garrapatas (Rhipicephalus) como vectores de transmisión biológica (Meli et al. 2010).
Hasta la fecha, sólo se ha reportado la existencia de dos especies en ganado bovino: M. wenyonii (Ayling et al. 2012) y Ca. M. haemobos (Tagawa et al. 2010). Ca. M. haemobos ya ha sido reportada en el ganado bovino de diferentes países causando efectos tales como anemia, depresión, edema, problemas reproductivos, mastitis, ictericia y pérdida de peso (Tagawa et al. 2010, 2012; Girotto et al. 2012; Hornok et al. 2012; Gladden et al. 2015; Martínez-Ocampo et al. 2016). Si bien no se ha observado un daño severo en los animales que la portan, la combinación de dos o más hemoplasmas (u otros patógenos) en un animal, compromete severamente su salud (Meli et al. 2010). Ca. M. haemobos podría ser considerada una bacteria “oportunista” ya que ha sido detectada en coinfecciones en el ganado bovino (Tagawa et al. 2013; Witter et al. 2017).
En los últimos años, este micoplasma se ha identificado en Suiza, Hungría, Alemania, China, Inglaterra, Nueva Zelanda y Japón, en tanto que en Latinoamérica sólo Brasil ha referido su presencia. En estos países se han desarrollado diferentes métodos moleculares para la detección de este micoplasma basados en PCR y qPCR (Meli et al. 2010; Nishizawa et al. 2010; Hoelzle et al. 2011; Ayling et al. 2012; Tagawa et al. 2012; McFadden et al. 2016).
Recientemente, se ha reportado la secuencia del genoma de Ca. M. haemobos identificado en México (Martínez-Ocampo et al. 2016), sin embargo, esta información no representa un estudio profundo de la presencia del micoplasma en el país. En el presente estudio, se logró detectar a Ca. M. haemobos mediante una prueba molecular, lo que representa un gran avance en el diagnóstico del patógeno y una aproximación del estudio de este hemoplasma, especialmente por su asociación a ciertos cuadros clínicos en combinación con otros microorganismos patógenos.
Por otro lado, se logró determinar la sensibilidad y especificidad de la prueba molecular. A su vez, se realizó un análisis bioinformático de las secuencias de los productos de PCR obtenidos en este trabajo y las secuencias de Ca. M. haemobos reportadas en otros países.
Materiales y métodos
Extracción de DNA genómico (gDNA) de sangre de bovinos
Se colectaron un total de 72 muestras de sangre: 11 muestras de Morelos (bovinos adultos ambos sexos), 12 de Durango (bovinos adultos machos), 9 de Veracruz (bovinos adultos ambos sexos), 14 de Jalisco (vaca-cría), 14 de Morelos (vaca-cría) y 12 de Querétaro (bovinos adultos hembras). Se tomaron alícuotas de 200 µL de las muestras de sangre seleccionadas.
Las muestras congeladas se colocaron en hielo hasta su descongelamiento y se extrajo el gDNA siguiendo el protocolo establecido en el UltraClean DNA Isolation Kit (MoBio, USA). El gDNA se eluyó en un volumen de 50 µL de agua de acuerdo con las indicaciones del protocolo. Para determinar la integridad del gDNA extraído, este fue sometido a una electroforesis en un gel de agarosa a 1% (p/v) con bromuro de etidio a 100 Volts por 40 minutos (Bio-Rad PowerPac 1000, Hercules, CA, USA). Después de la electroforesis, el gel fue documentado (EC3 Imaging System-UVP, LLC, Upland, CA, USA). La concentración del gDNA fue cuantificada por espectrofotometría (Nanodrop, ThermoFisher, Delaware, USA). Una vez cuantificadas las muestras, estas fueron utilizadas inmediatamente para PCR o almacenadas a -20°C hasta su uso.
Reacción en Cadena de la Polimerasa (PCR) de una región del gen 16S rRNA
Se realizó la amplificación por PCR punto final de la región comprendida entre los nucleótidos 109 y 605 del gen 16S rRNA de Ca. M. haemobos utilizando los oligonucleótidos (Mhfwd: 5’-ATC TAA CAT GCC CCT CTG TA-3’/Mhrev: 5’- GTA TTC GGT GCA AAC AA-3’) previamente reportados por Girotto et al. (2012). La reacción de PCR contenía 1 µL de cada oligonucleótido (10 pmol/ µL), 12.5 µL de MyTaq Mix 2X (Bioline, USA) y 300 ng de gDNA como templado en un volumen de reacción total de 25 µL.
Las condiciones de amplificación de la reacción de PCR fueron: 1 ciclo de desnaturalización inicial a 94°C por 3 minutos, posteriormente se realizaron 36 ciclos de 94°C por 30 segundos (desnaturalización), 58°C por 2 minutos (alineamiento), 72°C por 1 minuto (extensión), seguida por un ciclo de extensión final de 72°C por 10 minutos. Como control negativo se utilizó agua MilliQ esterilizada como templado y como control positivo se utilizó gDNA de una muestra de sangre infectada proveniente de Tizimín, Yucatán. Los productos de PCR (496 pb) fueron visualizados en un gel de agarosa a 1% teñido con bromuro de etidio. El gel se sometió a una electroforesis (Bio-Rad PowerPac 1000, Hercules, CA, USA) a 100 Volts por 40 minutos y fue documentado (EC3 Imaging System-UVP, LLC, Upland, CA, USA). Estas condiciones se utilizaron durante todas las reacciones de PCR realizadas para la detección de Ca. M. haemobos, secuenciación y pruebas de sensibilidad y especificidad. De cada reacción de PCR se realizaron tres repeticiones.
Purificación de los productos de PCR y preparación de las muestras para secuenciación
Se seleccionaron las muestras positivas a Ca. M. haemobos en los lotes de cada uno de los estados: 4D (Durango), 2G (Jalisco), 5M (Morelos), 12Q (Querétaro), 6M (Morelos, vaca-cría) y 3C (Morelos, vaca-cría), Se amplificó un producto de PCR de 496 pb del gen 16S rRNA con la finalidad de purificar el producto amplificado y secuenciarlo. Se cortó la banda del gel de agarosa en donde se encontraban los productos de PCR y después se purificaron con el kit Wizard Gel and PCR Clean-Up System (Promega, Wisconsin, USA). Los productos de PCR se eluyeron en un volumen final de 30 µL de agua libre de nucleasas y se almacenaron a -20°C. Los productos de PCR purificados fueron visualizados en un gel de agarosa a 1% teñido con bromuro de etidio. Con el objetivo de verificar la integridad del gDNA purificado, este fue analizado después de una electroforesis (Bio-Rad PowerPac 1000, Hercules, CA, USA) a 100 Volts por 40 minutos y documentado (EC3 Imaging System-UVP, LLC, Upland, CA, USA). Una vez purificado el producto de PCR se prepararon las muestras para su secuenciación. Para esto, se utilizaron tubos Eppendorf de tapa plana de 0.2 mL que contenían una concentración aproximada de 100-120 ng de cada producto purificado y 10 pmol de cada uno de los oligonucleótidos, posteriormente se ajustó todo a un volumen final de 16 µL. Las muestras fueron secuenciadas por ambos sentidos con los oligonucleótidos Mhfwd y Mhrev con el objetivo de ensamblarlas y verificar su identidad mediante un análisis en NCBI-Nucleotide BLAST. Las muestras fueron secuenciadas en la Unidad de Síntesis y Secuenciación del Instituto de Biotecnología de la UNAM.
Pruebas de sensibilidad y especificidad de la prueba de PCR
Las pruebas de sensibilidad se realizaron utilizando como templado las muestras que resultaron positivas para Ca. M. haemobos. Con base en los resultados de la cuantificación (Nanodrop, ThermoFisher, Delaware, USA) de los DNA genómicos extraídos, se ajustó la concentración de templado a 300 ng, 30 ng/ µL y 3 ng/µL, mediante las diluciones seriadas 1:10 y 1:100. Las condiciones de reacción y condiciones de la amplificación por PCR fueron las mismas descritas anteriormente. La prueba de especificidad se realizó utilizando como templado gDNA de Mycoplasma haemocanis. Esta muestra fue facilitada por la Unidad de Babesia del CENID-PAVET, INIFAP, cuantificada (Nanodrop, ThermoFisher, Delaware, USA) y ajustada a una concentración de 300 ng/µL para usarla como templado para la reacción de PCR con las condiciones mencionadas anteriormente. Los productos de PCR fueron visualizados en un gel de agarosa a 1% teñido con bromuro de etidio y sometido a una electroforesis a 100 Volts por 40 minutos. (Bio-Rad PowerPac 1000, Hercules, CA, USA) y documentado (EC3 Imaging System-UVP, LLC, Upland, CA, USA).
Alineamiento múltiple del gen 16S RNA
Se obtuvo le secuencia del gen 16S rRNA del hemoplasma Ca. M. haemobos INIFAP01 reportado en México (Martínez-Ocampo et al. 2016) utilizando el servidor RNAmmer (http://www.cbs.dtu.dk/services/ RNAmmer versión 1.2). Esta secuencia fue alineada con las secuencias 6Mor, 5Mor, 3C, 4D, 2G y 12Q y con 11 secuencias del gen 16S rRNA de hemoplasmas reportados en la base de datos de NCBI, ya sea como parte de genomas secuenciados o reportados individualmente como secuencia del gen 16S rRNA. Las secuencias se obtuvieron de la base de datos de GenBank (colección de nucleótidos [nr/nt]) utilizando el programa Nucleotide BLAST (Blastn). Se alinearon un total de 18 secuencias de genes 16S rRNA con el programa MUSCLE (https://www.ebi.ac.uk/Tools/ msa/muscle, versión 3.8.31).
Reconstrucción filogenética
La secuencia del gen 16S rRNA de Ca. M. haemobos INIFAP01 fue comparada con las secuencias 6Mor, 5Mor, 3C, 4D, 2G y 12Q y con 36 secuencias del gen 16S rRNA de hemoplasmas reportados, ya sea que estos hubieren sido referidos como parte de genomas secuenciados o individualmente como secuencia del gen 16S rRNA. Como grupo externo se utilizaron secuencias del gen 16S rRNA del género Ureaplasma. Las secuencias se obtuvieron de la base de datos de GenBank (colección de nucleótidos [nr/nt]) utilizando el programa Nucleotide BLAST (Blastn, https:// blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE_TYPE=- BlastSearch).
Un total de 44 secuencias de genes 16S rRNA se alinearon con el Programa MUSCLE (https:// www.ebi.ac.uk/Tools/msa/muscle versión 3.8.31). La selección estadística del mejor ajuste de modelos de sustitución de nucleótidos fue realizada con el programa JModelTest (http://evomics.org/learning/ phylogenetics/jmodeltest versión 2.1.10), se utilizó el modelo GTR+I+G. El análisis filogenético se realizó con el programa PhyML (http://www.atgc-montpellier.fr/ phyml versión 3.1) utilizando el método estadístico de máxima verosimilitud (ML por sus siglas en inglés “maximum likelihood”) con 1000 repeticiones de bootstrap. El árbol filogenético fue editado con el programa FigTree (http://tree.bio.ed.ac.uk/software/ figtree versión 1.4.3).
Resultados y discusión
Detección de Ca. M. haemobos en ganado bovino
Se realizó la amplificación por PCR de un producto de 496 pb que corresponde a la región del nucleótido 109 al 605 del gen 16S rRNA del hemoplasma Ca. M. haemobos. En todas las reacciones de PCR, se utilizaron 300 ng de gDNA como templado y en las pruebas de sensibilidad se utilizaron 30 y 3 ng como templado. De los lotes analizados se obtuvieron 18 muestras positivas de un total de 72, lo que representa 25% de muestras positivas para Ca. M. haemobos. Los porcentajes de positividad a Ca. M. haemobos son 9.09% en Morelos (1/11, muestra 5M), 50% en Durango (6/12, muestras 1D, 3D, 4D, 8D, 9D y 12D), 14.28% en Jalisco (vaca-cría) (2/14, muestras 1G y 2G) y 64.28% en Morelos (vaca-cría) (9/14, muestras 1CM, 2CM, 3M, 3CM, 6M, 6CM, 8M, 8CM, Y 9M). Tanto en las muestras de Querétaro (8Q es considerado un producto inespecífico dado su tamaño y los resultados de su secuenciación) como de Veracruz no se detectó Ca. M. haemobos (figura 1). La secuenciación de los productos de PCR confirmó que el producto amplificado corresponde a una región de la secuencia del gen 16S rRNA de Ca. M. haemobos, lo que evidencia que el uso de los métodos moleculares como PCR y la secuenciación son una herramienta útil para la detección e identificación de hemoplasmas (Meli et al. 2010; Nishizawa et al. 2010; Girotto et al. 2012; Hampel et al. 2014; Fu et al. 2017). Los resultados señalan que la positividad de las muestras es independiente del sistema de producción, dado que el porcentaje de positividad para Ca. M. haemobos varía entre las muestras analizadas provenientes ya sea de sistemas de confinamiento (Durango, Morelos) o de producción vaca-cría en pastoreo (Jalisco, Morelos).
Figura 1. Productos de PCR (496 pb) que amplifican una región del gen 16S rRNA de Ca. M. haemobos. Se obtuvieron muestras positivas a Ca. M. haemobos en muestras de Morelos, Durango, Jalisco. En muestras de Querétaro y Veracruz no se detectó al hemoplasma. C+ (control positivo), marcador de peso molecular (MPM). Los nombres de las muestras en color gris corresponden a muestras positivas por PCR.
Los porcentajes de las muestras analizadas positivas para Ca. M. haemobos son semejantes a los resultados de estudios referidos previamente en los cuales se estudió la prevalencia de M. wenyonii y Ca. M. haemobos en 109 muestras y se obtuvo que 12.8% resultaron positivas para ambos hemoplasmas (Nishizawa et al. 2010), mientras que otro estudio en Japón reportó que 5.1% de un total de 78 muestras resultaron positivas para M. wenyonii y Ca. M. haemobos (Tagawa et al. 2008).
Por otro lado, se realizó una comparación de la prevalencia de Ca. M. haemobos en ganado bovino de diferentes rangos de edades, con el objetivo de determinar si existe mayor susceptibilidad a patógenos en determinado rango de edad. Se agruparon las muestras en cuatro rangos de edad: 0-7 meses (14 muestras), 8-15 (13 muestras) y 16-36 (14 muestras) y > 37 (25 muestras). Se observó que en los lotes de bovinos analizados en un rango de 8-15 meses hay una mayor prevalencia de Ca. M. haemobos (46.1%); mientras que los bovinos en un rango de 0-7 meses tuvieron una prevalencia de 28.6%, seguidos de los bovinos de 15-36 meses con 21.4% y de los bovinos adultos de 37 meses con 16%. Los resultados muestran que los terneros analizados en un rango de 0-7 meses presentaron una menor prevalencia de Ca. M. haemobos, respecto a los terneros (8-15 meses).
A pesar de que se considera que los animales de 0-7 meses poseen la protección por anticuerpos maternos, es posible que la presencia de Ca. M. haemobos en estos animales sea consecuencia de una infección transmitida por vía intrauterina, como se ha observado en otras especies como Mycoplasma haemosuis,M. haemolamae, M. wenyonii y Ca. M. haemobos (Berrier y Gouge 1954; Meli et al. 2010; Hornok et al. 2012). El destete tradicional de bovinos se produce a los 7 meses (Reinhardt y Reinhardt 1981), por lo que es posible que los bovinos en etapa de desarrollo (8-15 meses) puedan haber estado en la etapa de destete, lo cual aumenta el estrés en los animales y los hace más susceptibles ante patógenos (Meli et al. 2010), además, estos pueden haber estado en etapa de vacunación, lo cual podría haber favorecido la trasmisión del hemoplasma por medio de fomites contaminados, como ocurre comúnmente en animales adultos (Hornok et al. 2011).
La prueba molecular utilizada en este estudio ha permitido realizar un estudio más profundo sobre la detección del hemoplasma Ca. M. haemobos en México, con lo que México se convierte en el segundo país latinoamericano, después de Brasil, en lograr su detección y reporte (Girotto et al. 2012). En cuanto a las pruebas de especificidad de la prueba de PCR utilizando muestras de gDNA de M. haemocanis y M. suis como templados de las reacciones de PCR, solamente se observó un producto de amplificación utilizando el gDNA de M. haemocanis. Este hecho concuerda con la reconstrucción filogenética de hemoplasmas que muestra la división de estos en dos clados y en donde se M. haemocanis se agrupa con Ca. M. haemobos (figura 2).
En lo que respecta a la sensibilidad de la prueba molecular, se observó la amplificación de productos de PCR utilizando 300 y 30 ng de templado, en tanto que empleando 3 ng no hay producto amplificado.
Analisis bioinformático
Se alinearon 18 secuencias del gen 16S rRNA de Ca. M. haemobos reportadas en la base de datos GenBank con el programa MUSCLE (versión 3.8). El alineamiento muestra porcentajes de identidad de entre 96.6% a 100%. En la figura 3, se muestra sólo la región alineada de las 18 secuencias en donde se observan su identidad, lo que sugiere que se trata de secuencias altamente conservadas.
Por otro lado, se realizó una reconstrucción filogenética basada en el gen 16S rRNA de las secuencias obtenidas de la secuenciación y en las secuencias reportadas en la base de datos GenBank. En la figura 2, se diferencian dos clados para las especies de hemoplasmas: clado A (azul) y B (rojo). El clado A está definido en los subclados AI y AII. En el subclado AI se agruparon los hemoplasmas que afectan al ganado bovino, incluyendo Ca. M. haemobos reportado en Japón, Suiza, Taiwán, Brasil y China, además de la cepa mexicana Ca. M. haemobos INIFAP01 y las secuenciadas en el presente estudio (6Mor, 5M, 3C, 4D, 2G, 12Q). En el subclado AII se agrupan los hemoplasmas que infectan a gatos y a perros (M. haemocanis Illinois, M. haemocanis Langford y M. haemofelis Ohio).
En el clado B, se agrupan especies de hemoplasmas que afectan una gran variedad de animales. En el subclado BI se agrupan animales como ratas y ratones (M. coccoides, Ca. M. haemomuris), monos (Ca. M. haemomacaque), humanos (Ca. M. haemohominis), gatos (Ca. M. turicensis), gatos y felinos salvajes (Ca. M. haemominutum), perros (Ca. M. haemoparvum), leones marinos (Ca. M. haemozalophi). En el subclado BII se agrupan especies de hemoplasmas que infectan a monos ardilla (Ca. M. kahanei), cérvidos (M. erythrodidelphis) y cerdos (M. suis y M. parvum). En el subclado BIII se agrupan aquellas especies de hemoplasmas que infectan llamas y alpacas (Ca. M. haemolamae), ciervos (Ca. M. erythrocervae), ganado bovino (Ca. M. haemocervae y M. wenyonii) y ovejas (M. ovis y Ca. M. haemovis).
Figura 2. Árbol filogenético de hemoplasmas basado en las secuencias del gen 16S rRNA. Los hemoplasmas se agrupan en el clado A (azul) y B (rojo). Los subclados se muestran los subclados AI y AII son del clado A, y BI, BII y BIII del clado B. El árbol filogenético fue construido con el programa PhyML (versión 3.1) utilizando el método estadístico de máxima verosimilitud con 1000 repeticiones de bootstrap. Las secuencias del género de Ureaplasma (grupo externo) se muestran en letras color verde. Los números de acceso de GenBank se muestran entre corchetes. En el rectángulo azul se muestra la cepa INIFAP01 (LWUJ01000012.1) y el círculo rojo señala las cepas utilizadas en este trabajo.
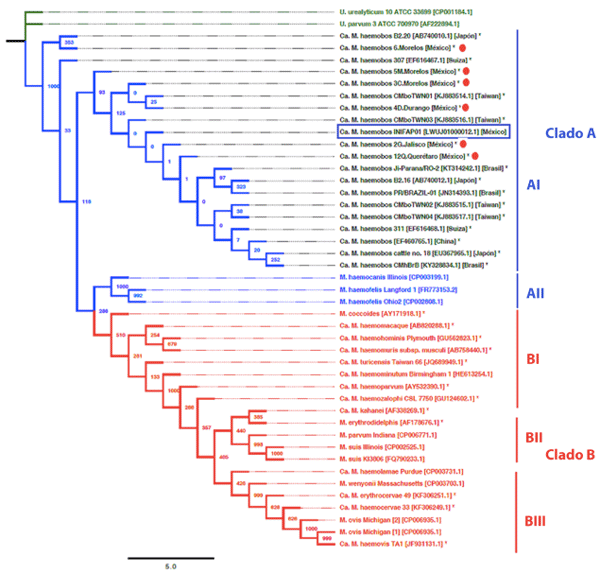
La reconstrucción filogenética basada en el gen 16S rRNA confirmó que las secuencias de los productos amplificados por PCR de una región del gen 16S rRNA corresponden efectivamente a Ca. M. haemobos. La filogenia muestra que existe una variabilidad del gen 16S rRNA entre diferentes especies de micoplasmas que infectan una gran variedad de animales. Por medio de análisis de genomas bacterianos se ha reportado que las especies clasificadas en un mismo clado como: Ca. M. haemobos, M. haemocanis y M. haemofelis (clado A) podrían estar compartiendo proteínas relacionadas con la patogenicidad, invasión y estructura. A pesar de que ambos clados están bien definidos, varios hemoplasmas que infectan al mismo tipo de hospedero están clasificados en clados diferentes (tal es el caso de Ca. M. haemobos y M. wenyonii), lo cual podría deberse a que se tratan de genomas altamente dinámicos, es decir, que presentan variaciones en el tamaño del genoma y mezclas de posiciones entre genes (Guimaraes et al. 2014).
Figura 3. Alineamiento de una región de las secuencias del gen 16S rRNA de Ca. M. haemobos. Se alinearon las secuencias de Ca. M. haemobos reportadas de acuerdo con el número de acceso de GenBank, Ca. M. haemobos INIFAP01 (LWUJ01000012.1, resaltada en color rojo) y las cepas de Ca. M. haemobos reportadas en este trabajo (6 Morelos, 3C Morelos, 12Q Querétaro, 4D Durango, 5M Morelos y 2G Jalisco). La identidad oscila entre 96 y 100%.
Conclusiones
Los programas de mejoramiento de la ganadería nacional enfrentan dificultades debido a las enfermedades transmitidas por hemoplasmas que afectan al ganado bovino, entre ellos Ca. M. haemobos. En este trabajo se ha implementado una prueba molecular para su detección evidenciado su presencia en 25% de las muestras analizadas. Por otro lado, los análisis filogenéticos muestran que la secuencia amplificada por PCR del gen 16S rRNA de Ca. M. haemobos se encuentra altamente conservada y que existen dos clados entre hemoplasmas que afectan diversos animales. La prueba molecular desarrollada permite identificar a los miembros del clado A, Ca. M. haemobos y M. haemocanis utilizando hasta 30 ng de DNA genómico extraído de sangre infectada de bovinos.












