Identificación de G tipos de rotavirus bovinos y porcinos encontrados en granjas porcinas y bovinas del Río Cuarto de Grecia
Publicado: 20 de marzo de 2007
Fuente: Bonilla José Luis, Bourdett Lurys y Jimenez Carlos / Universidad Nacional UNA, Costa Rica
Rotavirus bovino y porcino son la causa más frecuente de gastroenteritis. Varios tipos han sido identificados basados en dos proteínas de superficie VP4 (P-tipo) y VP7 (G-tipo). Sin embargo, estudios limitados han sido realizados en países tropicales como Costa Rica y otros de Centroamérica en identificar las cepas predominantes en porcinos y bovinos, debido probablemente a la disponibilidad de métodos apropiados. En este estudio se describe un método Reverso Transcriptasa-Reacción en Cadena de la Polimerasa (RT-PCR) para la tipificación de G-tipos para rotavirus porcinos y bovinos. Este método permitió identificar G-tipos de aislados colectados en granjas porcinas y bovinas del Río Cuarto de Grecia. Dentro de estos aislados se logró identificar G5 y G6 en lechones y terneros, respectivamente. Este estudio demostró que un método RT-PCR typing, es apropiado para estudios de la epidemiología molecular y que puede haber mayor diversidad entre rotavirus porcino y bovino que la previamente reportada. Palabras claves: Rotavirus porcino, Rotavirus Bovino, Diarrea, RT-PCR.
Introducción
Los rotavirus grupo A son una de las principales causas de enfermedades diarreicas en niños y animales jóvenes de todo el mundo (Paul et al. 1992; Saif et al. 1994; Kapikian et al. 1996). En granjas porcinas y bovinas las infecciones por rotavirus usualmente ocurren como afectación enzoótica afectando a más del 60% de la población de lechones y terneros (Paul et al. 1992; Saif et al. 1994). La mayoría de las infecciones naturales con rotavirus son subclínicas o están asociadas a diarreas moderadas. Sin embargo, muchas infecciones están asociadas con gastroenteritis moderada o severa (Woode, 1986; Paul et al. 1992).
Los rotavirus son clasificados dentro de la familia Reoviridae y son caracterizados por la presencia de una triple cápside viral y el genoma viral está compuesto por 11 segmentos de ARN doble cadena (Mattion et al. 1994). Basados en la especificidad de la VP6 localizada en la cápside interna del virus, los rotavirus pueden ser clasificados dentro de siete serogrupos distintos, denominados A, B, C, D, E, F y G. Las dos proteínas de superficie externa, VP4 y VP7, son responsables para activar anticuerpos de neutralización contra el virus. Un sistema binario es usado para clasificar los rotavirus del grupo A basados en la VP4 (P-tipos) y VP7 (G-tipos) específicamente (Estes et al. 1989). Estudios limitados han sido realizados sobre la prevalencia y distribución de serotipos de rotavirus porcinos y bovinos. En la actualidad las cepas que pertenece al grupo A de rotavirus porcino corresponden al serotipo G son 1, 3, 4, 5, 9, 10 y 11 (Bohl et al. 1984; Nagesha et al. 1988; Ciarlet et al. 1994; Pongsuwanna et al. 1996; Rach et al. 2000) han sido detectados. Dentro de los RV grupo A que circulan en bovinos se han reportado los G-tipos: G1, G2, G3, G6, G8, G10 y G11 (Gouvea et al. 1994; Falcone et al. 1999). Este estudio reporta el desarrollo de un método multiplex RT-PCR para tipificar P y G tipos y obtener un mejor conocimiento de la epidemiología molecular de rotavirus en porcinos y bovinos.
Material y Método
El estudio se realizó en una colección de 11 muestras en lechones a una edad aproximada de 1 día a 1 mes de vida, y 8 muestras de terneros de 1 día a 6 meses de edad, para un total de 19 muestras tomadas de granjas porcinas y bovinas del Río Cuarto de Grecia con presencia de diarrea. Las muestras fueron analizadas por la prueba Inmunoenzimática ligada a una enzima (ELISA) dirigido contra la proteína VP7, para rotavirus grupo A, empleando el ensayo IDEIA de Dako. Este kit comercial, utiliza un anticuerpo policlonal en un enzimoinmunoensayo “sandwich” de fase sólida, para la proteína VP7 (capside interna). Se sigue el protocolo de la casa comercial.
Genotipificación de para G (VP7) mediante RT-PCR
Extracción de ARN. Purificación de ARN de rotavirus. La extracción de muestras fecales fue realizada con Trizol LS (Gibco BRL# 10296-028) de acuerdo con el protocolo de la casa comercial. El botón que contiene el ARN se resuspende en 50μl de agua DEPC (0.1% diethyl pyrocarbonato, SIGMA) se guarda a -80°C hasta su posterior análisis.
Reverso Transcripción y Reacción en Cadena de la Polimerasa. Se basó en tres aspectos: la desnaturalización del dsRNA extraído, la transcripción reversa, utilizando la Transcriptasa Reversa, la amplificación del gen “G” (VP7), con los“primers” Beg9-End9 (Gouvea et al., 1994) y la genotipificación con el coctel de primers G5, G6, G8 y G10 (Gouvea et al., 1994) siguiendo la técnica descrita por Gouvea et al., (1990) y Gouvea et al (1994).
Tabla 1. Primers usados para RT-PCR de rotavirus porcino y bovino
Introducción
Los rotavirus grupo A son una de las principales causas de enfermedades diarreicas en niños y animales jóvenes de todo el mundo (Paul et al. 1992; Saif et al. 1994; Kapikian et al. 1996). En granjas porcinas y bovinas las infecciones por rotavirus usualmente ocurren como afectación enzoótica afectando a más del 60% de la población de lechones y terneros (Paul et al. 1992; Saif et al. 1994). La mayoría de las infecciones naturales con rotavirus son subclínicas o están asociadas a diarreas moderadas. Sin embargo, muchas infecciones están asociadas con gastroenteritis moderada o severa (Woode, 1986; Paul et al. 1992).
Los rotavirus son clasificados dentro de la familia Reoviridae y son caracterizados por la presencia de una triple cápside viral y el genoma viral está compuesto por 11 segmentos de ARN doble cadena (Mattion et al. 1994). Basados en la especificidad de la VP6 localizada en la cápside interna del virus, los rotavirus pueden ser clasificados dentro de siete serogrupos distintos, denominados A, B, C, D, E, F y G. Las dos proteínas de superficie externa, VP4 y VP7, son responsables para activar anticuerpos de neutralización contra el virus. Un sistema binario es usado para clasificar los rotavirus del grupo A basados en la VP4 (P-tipos) y VP7 (G-tipos) específicamente (Estes et al. 1989). Estudios limitados han sido realizados sobre la prevalencia y distribución de serotipos de rotavirus porcinos y bovinos. En la actualidad las cepas que pertenece al grupo A de rotavirus porcino corresponden al serotipo G son 1, 3, 4, 5, 9, 10 y 11 (Bohl et al. 1984; Nagesha et al. 1988; Ciarlet et al. 1994; Pongsuwanna et al. 1996; Rach et al. 2000) han sido detectados. Dentro de los RV grupo A que circulan en bovinos se han reportado los G-tipos: G1, G2, G3, G6, G8, G10 y G11 (Gouvea et al. 1994; Falcone et al. 1999). Este estudio reporta el desarrollo de un método multiplex RT-PCR para tipificar P y G tipos y obtener un mejor conocimiento de la epidemiología molecular de rotavirus en porcinos y bovinos.
Material y Método
El estudio se realizó en una colección de 11 muestras en lechones a una edad aproximada de 1 día a 1 mes de vida, y 8 muestras de terneros de 1 día a 6 meses de edad, para un total de 19 muestras tomadas de granjas porcinas y bovinas del Río Cuarto de Grecia con presencia de diarrea. Las muestras fueron analizadas por la prueba Inmunoenzimática ligada a una enzima (ELISA) dirigido contra la proteína VP7, para rotavirus grupo A, empleando el ensayo IDEIA de Dako. Este kit comercial, utiliza un anticuerpo policlonal en un enzimoinmunoensayo “sandwich” de fase sólida, para la proteína VP7 (capside interna). Se sigue el protocolo de la casa comercial.
Genotipificación de para G (VP7) mediante RT-PCR
Extracción de ARN. Purificación de ARN de rotavirus. La extracción de muestras fecales fue realizada con Trizol LS (Gibco BRL# 10296-028) de acuerdo con el protocolo de la casa comercial. El botón que contiene el ARN se resuspende en 50μl de agua DEPC (0.1% diethyl pyrocarbonato, SIGMA) se guarda a -80°C hasta su posterior análisis.
Reverso Transcripción y Reacción en Cadena de la Polimerasa. Se basó en tres aspectos: la desnaturalización del dsRNA extraído, la transcripción reversa, utilizando la Transcriptasa Reversa, la amplificación del gen “G” (VP7), con los“primers” Beg9-End9 (Gouvea et al., 1994) y la genotipificación con el coctel de primers G5, G6, G8 y G10 (Gouvea et al., 1994) siguiendo la técnica descrita por Gouvea et al., (1990) y Gouvea et al (1994).
Tabla 1. Primers usados para RT-PCR de rotavirus porcino y bovino
| Primer | 5´ a 3´ | cita |
| Beg9 | GGCTTTAAAAGAGAGAATTTC | Gouvea et al., 1994 |
| End9(UK) | GGTCACATCATACAACTCTAATCT | Gouvea et al., 1993 |
| End9(CRW8) | GGTCACATCTTACAGCTTTAACCT | Gouvea et al., 1993 |
| G5 (FT5) | CATGTACTCGTTGTTACGTC | Gouvea et al., 1994 |
| G6 (DT6) | CTAGTTCCTGTGTAGAATC | Gouvea et al., 1994 |
| G8 (HT8) | CGGTTCCGGATTAGACAC | Gouvea et al., 1994 |
| G10 (ET10) | TTCAGCCGTTGCGACTTC | Gouvea et al., 1994 |
Resultados
De un total de 19 muestras colectadas en bovinos y porcinos, se identificaron 5 muestras positivas en lechones y 6 muestras positivas en terneros mediante la prueba de ELISA.
Todas las muestras positivas al ELISA, fueron caracterizadas mediante la técnica de RT-PCR, para el caso de los terneros se detectó la presencia del genotipo G6 con un peso molecular de aproximadamente 500 bp y para el caso de los lechones el genotipo detectado fue G5, con un peso aproximado de 780 bp (Fig. 1).
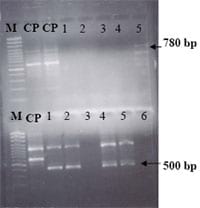
Figura 1. Representa los G tipos de rotavirus porcino (arriba) y bovinos (abajo), correspondientes a G5 (780 bp) y G6 (500 bp), respectivamente, utilizando un marcador de 100 bp.
Discusión
La especificidad de los serotipos de los aislados de campo y, consecuentemente, la disponibilidad de herramientas diagnósticas fiables hoy en día llegan a ser la relevancia primaria para el desarrollo de sistemas apropiados de vigilancia y control epidemiológico de infecciones por rotavirus porcino y bovino. En este contexto, las limitaciones de pruebas serológicas existentes son también reconocidas, mientras que las técnicas moleculares, tal como las prueba de PCR, ha quedado ampliamente aceptada como la prueba de elección para la rápida y completa caracterización de aislados de campo (Hussein et al. 1996).
En este estudio se detecta por primera vez la presencia del genotipo G6 en terneros, el cual es considerado el más prevalente en un estudio realizado en Argentina, donde mostró una presencia de 60% sobre el resto de los serotipos identificados (Garaicoechea L. et al. 2006). Otro estudio realizado en Ireland, también describe a G6 como el serotipo más prevalente con un 80% sobre resto (Reidy et al. 2006). Estudios realizados en Brasil muetran a G6 como el serotipo prevalente en un 42% con respecto de los otros encontrados (Fernández A. et al. 2004). Estos resultados se obtuvieron de terneros menores de 6 meses de edad con presencia de diarrea.
En el caso de rotavirus porcino el serotipo G5 se encuentra entre uno de los más comunes identificados según un estudio realizado en Brasil en cerdos de uno a sesenta días de edad con y sin presencia de diarrea, al cual también lo implican con posible rearreglo con serotipos P de humanos (Rácz M. et al. 2000); así también en un estudio realizado en Argentina mostró a G5 como el principal serotipo identificado en cerdos de uno a cuarenta y cinco días de edad con presencia de diarreas (Mattion et al. 1989).
La exactitud del análisis del serogrupo G realizado también resultó en un número muy bajo de cepas identificadas por la cantidad pequeña de muestras colectadas, por lo que deja en claro que puede haber mayor diversidad entre rotavirus porcino y bovino que la previamente reportada; por lo que en la medida de lo posible sería de mucha utilidad continuar con el estudio para obtener datos más significativos; ya que una de las ventajas de la clasificación y definición de los diferentes serogrupos de rotavirus porcino y bovino es la posibilidad de elaborar vacunas y entender más sobre la epidemiología molecular del virus ya que se ha considerado como potencial zoonótico (Palombo, 2002).
Agradecimiento
- A MSc. Lurys Bourdett, por su colaboración en el análisis y procesamiento de las muestras.
- Al DAAD por brindar el apoyo económico en la Maestría. A NeTropica y el CYTED, el cual son las instituciones financiadoras del proyecto.
- Al Dpto. de Virología de la Universidad Nacional, por poner a la disposición sus laboratorios.
Bibliografía
Paul, P.S., and G.W. Stevenson, 1992: Porcine rotavirus infection: Diseases of swine, 7th edition.Leman et al. (ed.), Iowa State University Press, Ames, IA, pp 331–348.
Saif, L. J., B. I. Rosen, and A.V. Parwani, 1994: Animal rotaviruses. In: A. Z., Kapikan, (ed.), Viral Infections of the Gastrointestinal Tract, 2nd edn, Marcel Dekker, Inc, New York, pp 335–367.
Kapikian, A.Z.and R. M.Chanock , 1996: Rotaviruses. In: Fields, B.N., D. M. Knipe and P. M. Howley (eds), Fields Virology, 3rd edn, pp.1657–1708 Lippincott-Raven Publishers, Philadelphia.
Woode, G. N., 1986: Porcine rotavirus infection. In: Leman (ed.), Diseases of Swine, 6th edn, pp.368–382. Iowa State University Press, Ames, IA.
Mattion, N.M. , J.Cohen, and M.K. Estes, 1994: The rotavirus proteins. In: A.Z.Kapikian , (ed.), Viral Infections of the Gastrointestinal Tract, 2nd Edn., Marcel Dekker, Inc., New York, pp.169–249.
Estes, M.K., and J. Cohen, 1989: Rotavirus gene structure and function. Microbiol. Rev. 53, 410–449.
Bohl, E.H. , K. W.Theil, and L.J.Saif, 1984: Isolation and serotyping of porcine rotaviruses and antigenic comparison with other rotaviruses. J.Clin.Microbi ol. 19, 105–111.
Nagesha, H.S. , and I.H.Holmes, 1988: New porcine rotavirus serotype antigenically related to human rotavirus serotype 3. J.Clin. Microbiol. 26, 171–174.
Ciarlet, M., M. Hidalgo, M. Gorziglia, and F. Liprandi, 1994: Characterization of neutralization epitopes on VP7 of serotype G11 porcine rotaviruses.J. Gen. Virol. 75, 1867–1873.
Pongsuwanna, Y., K. Taniguchi, M. Chiwakul, T. Urasawa, F. Wakasugi, C.Jayavasu and S.Urasawa, 1996: Serological and genomic characterization of porcine rotaviruses in Thailand: detection of a G10 porcine rotavirus.J. Clin.Microbiol. 34, 1050–1057.
Rach, M.L., S.S. Kroeff, V.Munfor d, T.A. R.Caruzo, E.L. Durigon, Y.Hayashi, V.Gouvea, and E.A Palombo, 2000: Molecular characterization of porcine rotaviruses from the southern region of Brazil: characterization of an atypical genotype G[9] strain.J. Clin.Microbiol, 38, 2443–2446.
Gouvea V., Santos N., and Timenetsky M., 1994, Identification of Bovine and Porcine Rotavirus G Types by PCR, Journal of Clinical Microbiology, 32(5): 1338-1340.
Falcone E., Tarantino M., L. Di Trani, Cordioli P., Lavazza A., y Tollis M, 1999. Determination of Bovine Rotavirus G and P Serotypes in Italy by PCR. Journal of clinical microbiology, 37:3879-3882.
Gentch, J.R., R. I.Glass, P.Woods, V.Gouvea, M.Gorziglia, J. Flores, B.K.Das, and M.K.Bhan, 1992: Identification of group A rotavirus gene 4 types by polymerase chain reaction. J. Clin. Microbiol. 30, 1365–1373.
Hussein, H. A., E. Frost, B. Talbot, M. Shalaby, E. Cornaglia, and Y. El-Azhary. 1996. Comparison of polymerase chain reaction and monoclonal antibodies for G-typing of group A bovine rotavirus directly from fecal material. Vet. Microbiol. 51:11–17.
Palombo E., 2002, Genetic of group A rotavirus: Evidence for Interspecies transmission of Rotavirus genes, Virus Genes, 24(1): 13-20.
Garaicoechea L., Bok K., Jones L. R., Combessies G., Odeón A., Fernandez F., and Parreño V., 2006, Molecular characterization of bovine rotavirus circulating in beef and dairy herds in Argentina during a 10-year period (1994–2003). Veterinary Microbiology, 118:1-11.
Reidy N., Lennon G., Fanning S., Power E., and O’Shea H., 2006, Molecular characterisation and analysis of bovine rotavirus strains circulating in Ireland 2002–2004, Veterinary microbiology, 117:242-247.
Fernandes A., Alcindo A., Bacellar M., Gagliardi Leite J., and Richtzenhain L., 2004. G and P genotypes of group A rotavirus strains circulating in calves in Brazil, 1996–1999, Veterinary Microbiology, 99:167-173.
Rácz M., Kroeff S., Munford V., Thabata A. R., Durigon E., Hayashi Y., Gouvea V., and Palombo E., Molecular Characterization of Porcine Rotaviruses from the Southern Region of Brazil: Characterization of an Atypical Genotype G[9] Strain, Journal of Clinical Microbiology, 38(6):2443-2446.
Mattion M., Bellinzoni R., Blackhall J., La Torre J., and Scodeller E., 1989, Antigenic Characterization of Swine Rotaviruses in Argentina, Journal of Clinical Microbiology, 27(4):795-798.
Temas relacionados:
Mencionados en esta noticia:
Recomendar
Comentar
Compartir

23 de noviembre de 2008
Dr. Jose Luis Bonilla:
Felicitaciones por su trabajo. Mi nombre es Luz Marina Barrera, soy estudiante de Maestria en Produccion Animal Tropical en la Universidad Nacional de Colombia, y mi proyecto de tesis es sobre un parasito equino Anaplocephala perfoliata (cestodo), se pretende utilizar la tecnica de PCR para copiar su ADN. Le agradeceria inmensamente si tiene datos sobre este parasito, y si es posible para mi obtener todo el trabajo que han realizado sobre los rotavirus.
Recomendar
Responder
8 de mayo de 2007
Quizá no me debiera entrometer, pero conozco del proyecto ya que algunos veterinarios de nuestra cooperativa aportaron muestras. Probablemente el tesiario es de León, Nicaragua, pero el lugar donde se realizó el estudio es Río Cuarto de Grecia, en Costa Rica.
El Dr. Carlos Jiménes es el director de la Escuela de Medicina Veterinaria de la UNIVERSIDAD NACIONAL DE COSTA RICA.
Gonzalo Carmona Solano
Recomendar
Responder
24 de noviembre de 2008
Hola Ing. Luz Marina Barrera, quería comunicarle que aun no estamos realizando trabajos sobre este parásito en especial si usted desea que le mande informacion sobre los trabajos que hemos realizado sobre rotavirus por favor mándeme su correo electrónico.
Gracias...
Saludos...
Recomendar
Responder

21 de noviembre de 2008
Dr. jose luis bonilla espinoza es una gusto comunicarme con usted mi nombre es miguel rojas bachiller de medicina veterinaria de perú hace unos meses le pedi informacion sobre rotavirus porcino usted accedió y me pidió el correo aquí le doy uno.
atakerasta84@hotmail.com me encantaría darle el de la universidad pero por motivo de ser egresado a nosotros nos anulan ese correo muchas gracias por la informacion q me pueda brindar .
atte miguel rojas.
Recomendar
Responder


Micotoxinas en cerdos. Diagnosis y Relación con enfermedades víricas
Enlace recomendado
19 de mayo de 2008
Claro que si con mucho gusto le puedo enviar información, si usted seria tan amable de enviarme su correo electrónico, le enviaré información.
Gracias...
Recomendar
Responder

18 de mayo de 2008
Dr jose luis bonilla es un placer comunicarme con usted mi nombre es miguel soy bachiller de medicina veterinaria de la UNMSM en lima peru, mas q un comentario me dirijo a usted como un pedido. Dr bonilla estoy trabajando en mi tesis la cual trata de rotavirus porcino estoy usando PAGE y queria saber si tiene informacion q pudiera compartir referente a los electroferotipos q se presenten en los porcinos. en si Dr cualquier informacion sobre rotavirus seria bienvenida le agradesco de ante mano su atencion.
atte
Recomendar
Responder
9 de mayo de 2007
Así es Dr. Carmona, yo soy la persona que está trabajando con el proyecto Rotavirus, tal parece que hubo un pequeño error, así es el estudio se está realizando en Costa Rica, supervisado por el Dr. Jiménez.
Ya le escribí a la revista para que se pueda corregir el pequeño error.
Recomendar
Responder
8 de mayo de 2007
Así es Dr, el Dr. Carlos Jiménez es el Director de la carrera de Medicina Veterinaria de la UNA, y el responsable del laboratorio de virología, donde se está trabajando para lograr caracterizar cuaáes son los serotipos de rotavirus porcino y bovino presentes en Costa Rica... Este fue un breve avance de lo encontrado hasta el momento.
Le agradezco de antemano su opinión.
Recomendar
Responder
7 de mayo de 2007
José Luis Bonilla, un saludo a la distancia y a la vez, le pido que me aclare si el Dr. Jiménez es el del departamento de Virología de la Escuela de Medicina Veterinaria, UNA, Heredia, Costa Rica. Y si el lugar mencionado, es Río Cuarto de Grecia, Alajuela, Costa Rica. ¿O me equivoco?
Por cierto, muy buen trabajo. Los Felicito. Importante aporte de información.
Gracias!
Recomendar
Responder


Inmunología porcina: lecciones para comprender mejor el SARS-CoV-2
Enlace recomendado

¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.











